引言
目前,阿尔茨海默病(Alzheimer's Disease,AD)在中国已有1 400万患者,预计到2050年,这个数字将超过3 000万[1].AD患者常常表现为记忆力衰退、思维能力下降、语言理解和表达能力减弱等症状,最终可能丧失自理能力.尽管目前尚无有效治疗AD的方法,但患者可以通过药物治疗、认知训练和运动来延缓疾病的进展.轻度认知障碍(Mild Cognitive Impairment,MCI)通常被认为是AD的早期阶段,表现为人体记忆能力的衰退.因此,准确及时地诊断AD和MCI对于减缓并干预AD发展进程至关重要.磁共振成像(Magnetic Resonance Imaging,MRI)已经成为神经系统疾病诊断的重要工具,特别是T1加权成像,它能够揭示大脑结构的异常情况,如灰质萎缩和脑区体积变化.T1加权成像在临床上对于区分AD与其他认知疾病具有重要作用[2,3].尽管T1加权成像包含了丰富的解剖信息,但临床医生通过影像阅片来诊断疾病仍然存在不少困难.为了提高诊断的准确性,很多研究使用基于体素的形态测量[4],基于张量的形态测量[5]和多视角形态特征[6]与机器学习算法相结合用于AD分类.这些方法减少了不同医生之间的诊断差异,同时也显著提高了AD的诊断水平.
然而以上方法在特征提取时依赖手工设计的特征,这不仅需要专业的知识,而且容易遗漏有用信息,因此在临床应用中缺乏一定的稳健性.近年来,作为机器学习的重要分支,深度学习凭借无需进行复杂的特征计算和较少预处理的优势,在医学影像领域的应用发展迅速,如医学影像分割[7]、检测[8]和分类[9].目前,深度学习在AD分类的应用主要集中于二维和三维图像的处理.Jain等[10]从三维结构磁共振成像(Structural Magnetic Resonance Imaging,sMRI)中精选出信息量丰富的2D切片作为输入,并将在ImageNet数据集上训练的权重迁移至VGG16网络中进行训练,结果显示在AD、MCI和正常对照(Normal Control,NC)的分类任务中取得了95.73%的准确率.Kang等[11]使用sMRI图像的冠状面切片在基于ImageNet迁移学习的VGG16模型中进行训练,根据AD和NC的分类效果选择分类精度最高的前11张冠状面图像,然后将这些图像输入到VGG16和ResNet50中进行进一步的训练,最后使用多数投票方案集成多个切片的预测结果.该方法对AD与NC、AD与MCI、MCI与NC的分类准确率分别为90.36%、77.19%、72.36%.虽然二维图像在AD分类中的应用已经取得一定的进展,但仍存在一些局限性.二维切片只能捕捉到局部信息,可能会忽略三维结构中重要的空间关系.如果选择特定的切片作为输入也有可能会导致信息丢失,而且不同个体的大脑解剖结构存在差异,单纯依赖二维切片进行分类可能会影响模型的泛化能力.为了克服这些局限性,利用三维图像进行AD分类成为一种更为全面和有效的方法.三维图像无需进行切片选择,能够保留完整的空间信息,使得模型可以更好地理解和捕捉脑部结构,从而提高分类的准确性和鲁棒性.Payan等[12]首次使用3D卷积神经网络对AD、MCI和NC进行分类,并实现了89.47%的准确率.Bakkouri等[13]在3D卷积神经网络的基础上进行改进,提出了一种多尺度卷积神经网络,在该网络的每个模块中采用多尺度卷积层,并将特征图在通道维度上进行拼接,从而增强了网络的多尺度特征提取能力.最终,其模型在AD与NC,AD与MCI和MCI与NC分类任务中准确率分别达到了97.03%、93.42%、95.90%.Yee等[14]提出了一种高效且轻量级的3D卷积神经网络,该网络采用空洞卷积增强捕捉多尺度信息的能力,在AD和NC分类中达到了88%的准确率.Yu等[15]将2D ResNet改为3D ResNet模型,该模型在AD和NC分类任务中取得了97.425%的准确率.虽然这些研究方法有一定的临床应用潜力,但也存在一些限制.例如,卷积注意力模块(Convolutional Block Attention Module,CBAM)[16]通过结合通道注意力和空间注意力已经取得了显著的成果,但也带来了额外的模型复杂度.同时,AD和MCI的病理性改变可能发生在全局或局部,并且在空间尺度和位置上存在变化.然而,现有方法在融合多尺度特征时,通常采用求和或通道拼接的方式,这些操作会引入冗余噪声,降低了特征表达的有效性,不利于模型的训练和性能的提高.
受上述研究工作的启发,本文提出了一种基于深度学习的三维分类模型(3D HAMSNet),该模型能够准确地区分AD、MCI和NC.本研究贡献具体如下:
(1)提出一个新的混合注意力机制,在通道注意力中引入了通道自适应注意力模块,使模型能够在训练过程中自适应地调整最大池化和平均池化策略,以便更精确地识别并聚焦于关键的通道,在空间注意力机制中,通过平均池化和最大池化提取空间特征,生成单通道特征图,并通过拼接和卷积生成空间注意力权重图,使模型聚焦于特征图中的关键区域,从而提升特征表达能力,提高模型在分类任务中的性能.
(2)提出一种新的多尺度信息融合模块,该模块在多尺度特征提取中,采用了具有不同扩张率的空洞卷积层来代替传统的多尺度卷积层,从而提高特征提取的效率.此外,在特征选择和融合阶段引入了软注意力机制,可以有效减少冗余尺度信息带来的噪声问题.
在本文后续内容安排中,先对所提出的方法进行了详细介绍,包括:整体流程图、网络结构、3D卷积、混合注意力模块和基于空洞卷积和软注意力的多尺度信息融合模块,接着对数据集构建和实验设置细节进行了说明,并通过消融实验和对比实验对所提出的方法进行了验证和结果展示,最后给出了本文的总结.
1 实验方法
1.1 整体流程
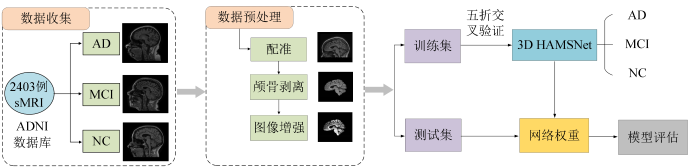
本文实验整体流程图如图1所示.首先,对数据集进行预处理操作,以确保数据的质量和适用性,并将数据集划分为训练集和测试集.接着,在训练阶段,采用五折交叉验证的策略进行模型训练,获得每个模型的权重.最后,使用测试集对训练好的模型进行评估,并获取最终的预测结果.
图1
1.2 网络结构
本实验在卷积神经网络(Convolutional Neural Networks,CNN)基础上进行改进.虽然CNN模型最初应用于2D图像,但MRI等3D医学图像通常包含丰富且重要的空间信息.相比于2D深度学习模型,3D深度学习模型能更好地识别MRI图像中的解剖结构.为了充分利用sMRI图像中的空间上下文信息,本文采用3D CNN代替2D CNN.3D CNN主要分为两个部分:特征提取层和分类层.特征提取层主要分为卷积层、激活函数与池化层,在分类层中,通过全连接层进行降维,最终得到分类结果.
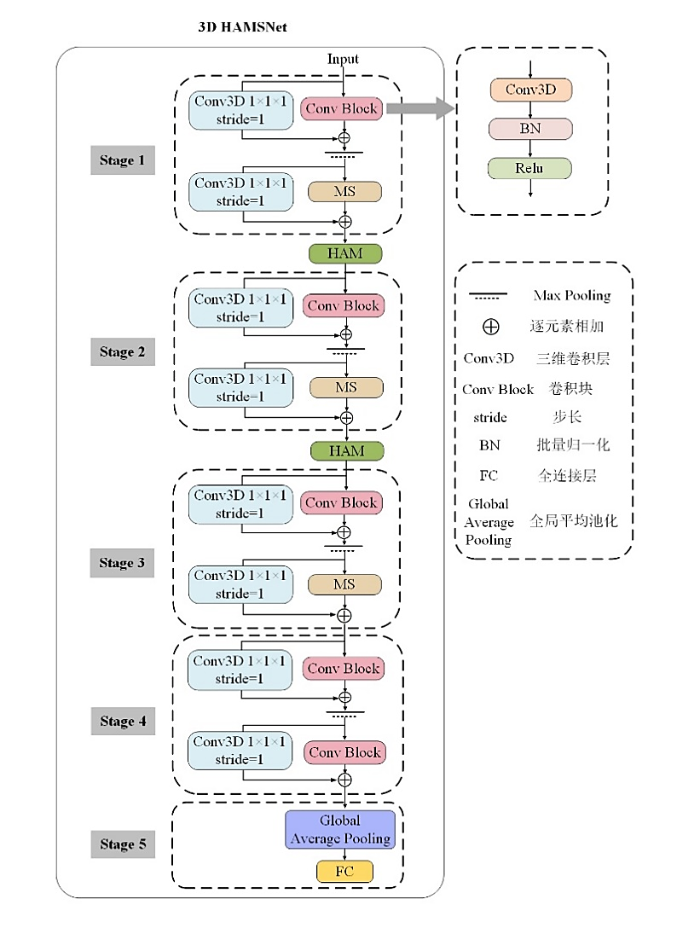
基于前述条件,我们提出一种3D HAMSNet模型,其网络结构图如图2所示.模型包括五个阶段,前三个阶段每个都由两个卷积层、一个卷积块和多尺度信息融合模块(Multiscale Information Fusion Module,MS)组成,其中卷积块包含卷积层,批量归一化以及ReLU激活函数,用于有效地提取浅层特征表示.第四个阶段包括两个卷积层和两个卷积块,以进一步提取高级语义信息.受ResNet[17]启发,在卷积块和多尺度信息融合模块中引入了残差连接,提高特征表示的学习能力.为了更好地捕捉上游输入的特征,在前两个阶段之后都添加了混合注意力模块(Hybrid Attention Module,HAM).最后,模型采用全局平均池化层和全连接层作为分类器用于最终分类.
图2
1.3 3D卷积
二维卷积神经网络在处理二维图像时表现出优异的效果,其构建的网络能够自动提取图像中的颜色、纹理和形状等特征.在本研究中,我们在二维卷积的基础上增加了一个维度,需要注意的是,这里的维度不是通道维度,而是指滑动卷积核在空间上的维度.3D卷积与2D卷积类似,具体公式如下:
其中O代表输入的3D数据,
1.4 混合注意力模块
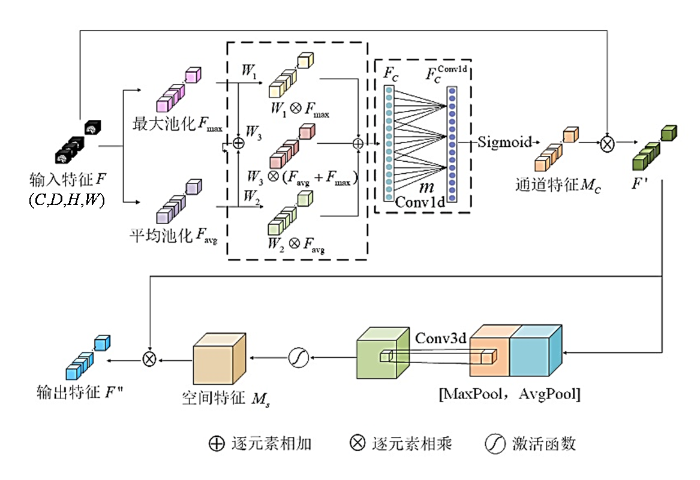
在AD和MCI的MRI分析中,普遍观察到患者全脑体积和结构的广泛性改变.但是,研究表明某些脑区,如海马体[18]、杏仁核[19]和颞叶[20]等,在病理变化上表现出更为明显的萎缩和变形,这些脑区的结构退行性变化与AD和MCI密切相关,并在疾病进程中发挥着关键的作用.因此,为了使网络关注通道和空间特征,在网络中添加了HAM.受压缩和激励(Squeeze-and-Excitation,SE)[21]模块和卷积块注意力模块(Convolutional Block Attention Module,CBAM)[16]的启发,考虑到通道关系的重要性,本文设计了通道自适应注意力模块.在SE模块中,通过平均池化对输入特征图的空间维度进行压缩.基于这一理念,Woo等[16]进一步证明最大池化也能提供重要的线索,因此,在CBAM的通道注意力机制模块中,分别对输入特征图进行空间维度的平均池化和最大池化,以聚合空间特征.然而,平均池化和最大池化在特征提取的不同阶段发挥着不同的作用.为了解决这一问题,在通道注意力模块中加入通道自适应模块,该模块能够在不同阶段动态地调整池化策略,使平均池化和最大池化在各自的阶段中发挥最佳效果.此外,使用自适应核大小的一维卷积避免通道的降维和降低模型的复杂性,并捕获跨通道的相互作用.
HAM的结构如图3所示,由通道注意力模块和空间注意力模块构成.在通道注意力模块中,输入特征图为
其中,
其中σ表示激活函数,
图3
在空间注意力模块中,输入特征
1.5 基于空洞卷积和软注意力的多尺度信息融合模块
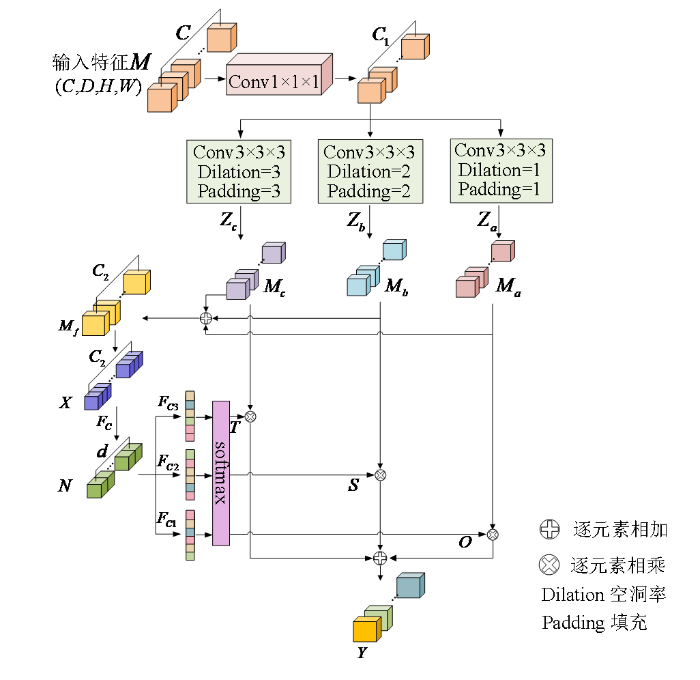
为了充分地利用sMRI图像的空间信息,提高特征提取的效率并减少特征融合时产生的噪声,本文提出了一种基于空洞卷积和软注意力的多尺度信息融合模块,其结构如图4所示.
图4
多尺度信息融合模块由特征提取、特征选择和特征融合三个部分组成.第一部分是特征提取,首先,将输入特征
然后,对融合后的特征图
其中,
其中,
第三部分是特征融合,分别使用三个独立的全连接层
其中,
多尺度融合特征图为
2 实验设置
2.1 实验数据集
本文研究利用的数据来自阿尔茨海默症神经影像倡议(Alzheimer's Disease Neuroimaging Initiative,ADNI)数据库,该数据库公开提供sMRI、基因数据以及其他生物标记和相关诊断信息,成为研究阿尔茨海默病的标准数据来源[24].本文实验数据是由ADNI数据库下载的537名受试者(198名AD,200名MCI和139名NC)的sMRI图像组成,每个受试者可能有多次扫描数据,共2 403例sMRI影像.
本文采用五折交叉验证的方法平衡训练和评估性能.为避免数据泄露,将数据按受试者随机分为五个子集,并确保来自同一受试者的sMRI影像只出现在同一个子集中.此分组方法保证了不同子集之间数据的独立性,从而提高了实验结果的可靠性和有效性.训练集和测试集的比例约为90%和10%,如表1所示.在训练过程中,将训练集均分为五份,每次选取其中一份作为验证集,其余四份作为训练集,重复五次.最终,模型性能通过测试集上的评估结果取平均值来衡量.
表1 实验数据的划分
Table 1
| 数据类型 | 训练集 | 测试集 | 总计 |
|---|---|---|---|
| AD | 721 | 79 | 800 |
| MCI | 724 | 80 | 804 |
| NC | 719 | 80 | 799 |
| 总计 | 2164 | 239 | 2403 |
2.2 数据预处理
原始脑部sMRI图像中可能含有一些非大脑结构,如头骨,这些结构可能会作为噪声干扰图像,影响实验结果.因此,在对数据进行网络模型的训练之前进行预处理是十分必要的.为此,许多研究采用FMRIB软件库(FSL,
首先,将原始数据与MNI152_T1_1mm模板进行配准对齐,使相似的结构对准到相同的空间位置,从而使得网络能更好地识别到相关特征.然后,使用FSL的脑提取工具(BET)从配准后的数据中去除所有非脑组织.最后,对配准后的图像进行中值滤波和直方图均衡化处理,以增强图像质量,并将图像大小裁剪为112×96×96.图5为预处理后的结果.
图5
图5
图像经过不同预处理步骤后的结果
Fig. 5
Results of images after different preprocessing steps
2.3 评估指标
为了验证所提出3D HAMSNet模型在AD、MCI和NC分类中的性能,本实验采用了五个常用的性能指标,即准确率(ACC)、特异性(SPE)、灵敏度/召回率(SEN)、精确率(PRE)以及F1分数(F1).计算公式如下:
公式中真阳性(TP)表示正样本的正确预测数,真阴性(TN)表示负样本的正确预测数,假阴性(FN)表示正样本的错误预测数,假阳性(FP)表示负样本的错误预测数.
2.4 参数设置
在本实验中,所提出的模型采用Adam优化器进行训练,初始学习率设置为0.000 2,每5个epoch衰减0.9.本文使用ResNet50模型进行实验,并在保证其他参数不变的条件下,对不同epoch数量进行了比较,如表2所示,模型的准确率并未随着epoch数量的增加而一直提高,当epoch数量达到40时,准确率最高.因此,本文选择将epochs设置为40,并将batch size设置为16.采用交叉熵损失函数作为实验的损失函数,计算公式如下所示:
其中,
表2 ResNet50在不同epoch数量下的准确率
Table 2
| epochs | ACC/(%) |
|---|---|
| 20 | 76.62 |
| 30 | 84.10 |
| 40 | 86.61 |
| 50 | 85.77 |
| 60 | 85.77 |
3 实验结果与分析
3.1 结果评估及消融实验
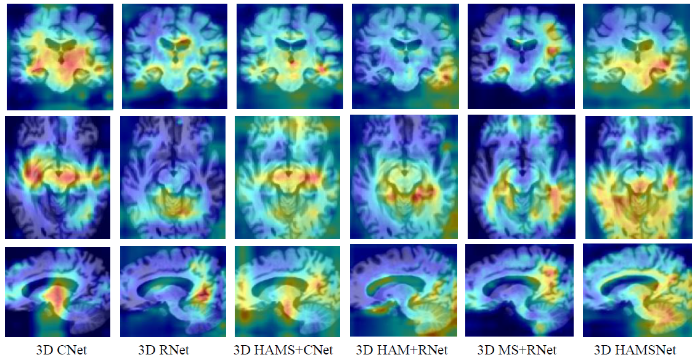
为验证模块的有效性,本文进行了消融实验,并通过五折交叉验证.实验结果如表3所示.3D CNet是一种不含混合注意力、多尺度信息融合模块和残差连接的网络,其特征提取主要依赖$3 \times 3 \times 3$卷积核的堆叠,尽管该网络设计简洁,并且能够在一定程度上捕捉图像的特征,但其分类准确率和F1分数在几种模型均最低.3D RNet在3D CNet的基础上引入了残差连接,残差连接提高了网络的特征传递能力和梯度反向传播效率,同时保持了网络的结构简洁,与3D CNet相比,准确率提升了3.19%.3D HAM+RNet通过在3D RNet中引入混合注意力机制,混合注意力机制的加入能够有效地选择关注重要的通道特征并结合空间信息,使得三分类准确率达到了93.39%,可见混合注意力机制在AD分类中起到了重要作用.3D MS+RNet是在3D RNet中添加多尺度信息融合模块,该模块有效地融合了AD的多种空间尺度特征,虽然准确率提升幅度不如3D HAM+RNet,但在一定程度上也提高了AD的整体性能.3D HAMS+CNet表示在3D CNet中添加了混合注意力机制和多尺度信息融合模块,相比于3D CNet,其分类准确率和F1分数分别提高了4.94%和5.27%,虽然引入了两个对模型有积极影响的模块,但组合使用后,准确率提高并不是最高的,原因可能在于未引入残差连接.从表3中可以看出,残差连接显著提高了模型的特征传递能力和梯度反向传播效率,因此3D HAMS+CNet在这些方面仍然存在不足,限制了其性能的进一步提升.3D HAMSNet是本文提出的网络模型,该模型表现最佳,准确率和F1分数分别为94.14%和94.17%,相较于3D CNet,准确率、特异性和F1分数分别提升了9.88%、4.94%和10.17%.图6通过混淆矩阵的形式展示了3D HAMSNet模型在五折交叉验证策略下对测试集进行分类的效果.表4展示了所提出3D HAMSNet在五折交叉验证中各个分类指标的具体数值.消融实验的结果表明本文提出模块可以提高AD、MCI和NC的分类效果.
表3 不同模块的消融实验结果表
Table 3
| 模型 | ACC/(%) | SPE/(%) | SEN/(%) | PRE/(%) | F1/(%) |
|---|---|---|---|---|---|
| 3D CNet | 84.26(±2.53) | 92.13 | 84.26 | 87.38 | 84.00 |
| 3D RNet | 87.45(±2.44) | 93.72 | 88.20 | 88.65 | 87.61 |
| 3D HAMS+CNet | 89.20(±1.53) | 94.60 | 89.21 | 89.84 | 89.27 |
| 3D HAM+RNet | 93.39(±1.31) | 96.70 | 93.39 | 93.46 | 93.42 |
| 3D MS+RNet | 91.73(±1.80) | 95.90 | 91.80 | 92.16 | 91.83 |
| 3D HAMSNet | 94.14(±1.03) | 97.07 | 94.14 | 94.22 | 94.17 |
图6
表4 五折交叉验证结果
Table 4
| 模型 | ACC/(%) | SPE/(%) | SEN/(%) | PRE/(%) | F1/(%) |
|---|---|---|---|---|---|
| 第一折 | 94.98 | 97.49 | 94.98 | 94.98 | 95.54 |
| 第二折 | 95.40 | 97.70 | 95.40 | 95.41 | 95.57 |
| 第三折 | 93.72 | 96.86 | 93.71 | 93.72 | 93.22 |
| 第四折 | 94.14 | 97.07 | 94.14 | 94.37 | 94.11 |
| 第五折 | 92.47 | 96.23 | 92.47 | 92.60 | 92.43 |
图7
3.2 对比实验
为验证本文提出方法的有效性,我们将3D HAMSNet与3D VGG16[27]、3D DensNet121[28]、3D ResNet34[17]、3D ResNet50与3D Vision Transformer(3D ViT)[29]进行对比.所有的比较实验均在本文数据集上进行,然后利用五折交叉验证,参数设置与2.4节一致.表5展示了各网络的分类性能.实验结果显示,我们提出的网络3D HAMSNet分类效果最佳,准确率为94.14%,灵敏度为94.14%,特异性为97.07%.由于3D_VGG16采用堆叠式方法进行特征提取,导致其在特征提取方面存在一定的局限性,因此分类准确率相对较低.尽管3D_DensNet121标准差低于3D_ResNet50,但其准确率低了9.89%.3D ViT采用自注意力机制捕捉长程依赖关系,导致其在特征提取时未能充分利用局部特征,所以影响了分类的准确率.在预测阶段,本实验提出的3D HAMSNet在测试集上更突出地展示了其在AD、MCI和NC的分类性能.
表5 对比实验结果
Table 5
| 模型 | ACC/(%) | SPE/(%) | SEN/(%) | PRE/(%) | F1/(%) |
|---|---|---|---|---|---|
| 3D_VGG16 | 69.29(±5.79) | 84.64 | 69.34 | 72.64 | 68.80 |
| 3D_DensNet121 | 74.56(±2.37) | 87.28 | 76.10 | 77.59 | 74.17 |
| 3D_ResNet34 | 83.26(±2.53) | 89.03 | 83.32 | 84.40 | 82.86 |
| 3D_ResNet50 | 87.45(±2.44) | 93.72 | 88.20 | 88.65 | 87.61 |
| 3D ViT | 69.93(±2.41) | 82.40 | 69.81 | 69.62 | 69.39 |
| 3D HAMSNet | 94.14(±1.03) | 97.07 | 94.14 | 94.22 | 94.17 |
4 讨论与总结
对于AD患者而言,快速准确地分类出AD、MCI和NC至关重要.然而,不同医生通过临床经验对诊断结果的判定还是存在差异,基于深度学习的辅助诊断可以有效减少这种差异,提高诊断的准确性和一致性.在本实验中,我们在CNN的基础上进行改进,引入了残差连接,混合注意力机制和多尺度信息融合模块,构建了一个能自动诊断AD、MCI和NC的深度学习网络(3D HAMSNet),在三分类任务中该模型的准确率为94.14%,灵敏度为94.14%,特异性为97.07%.与以往研究相比,Bakkouri等[13]提出的基于3D多尺度特征块和门控循环融合单元的模型,通过使用多尺度特征块,能够有效提取不同分辨率的特征信息,从而增强对大脑结构细微变化的识别能力.然而,该模型在通道和空间注意力的关注上较为不足,可能影响特征选择的精准性和对海马体、杏仁核和颞叶等重要区域的关注.Tong等[30]提出的基于3D CNN的空间上下文网络通过融合3D CNN提取的特征与原始MRI图像的空间信息,有效捕捉MRI切片间的相关特征,虽然在AD与NC分类中表现良好,但在区分MCI与AD和NC时相对较弱,这可能是因为模型对边缘系统和脑部细微结构变化的关注不足.
本文提出的3D HAMSNet模型相较于以往研究进行了改进,首先,将2D CNN修改为3D CNN,以更好地捕捉sMRI图像中的空间上下文信息,并在卷积块和多尺度信息融合模块中引入残差连接,从而在保证模型结构简洁的同时提高了特征传递能力和梯度反向传播效率.针对模型对海马体、杏仁核和颞叶等区域的关注问题,引入了改进的混合注意力机制,在CBAM的通道注意力模块中引入了通道自适应注意力模块,以动态调整各通道的特征权重.通过使用自适应核大小的一维卷积代替多层感知器,模型能避免通道降维带来的负面影响,同时降低了模型的复杂性.同时,采用基于空洞卷积和软注意力的多尺度信息融合网络,有效提取多尺度感受野信息,捕获AD患者脑部萎缩可能发生的脑区域,进一步提高特征提取的效率并减少计算负载.然而,本实验还存在一些不足之处.首先,本研究仅使用了T1加权图像进行分析,未来可以考虑采用多模态的方法以提高诊断的准确率.其次,本文仅对AD、MCI和NC进行分类,而MCI又可以进一步分为进展型轻度认知障碍和稳定型轻度认知障碍,未来的研究可以根据疾病的发展趋势对MCI进行更细致的分类以更好地了解疾病的发展趋势.最后,随着年龄的增长,脑组织的结构变化可能会加剧,可能会导致脑萎缩的程度加深,其他退行性疾病也会对脑萎缩产生影响,在未来研究中可以将这些因素加入模型中,以提高分类的准确性.综上所述,本研究提出的3D HAMSNet模型在辅助诊断阿尔茨海默病方面展示了良好的潜力,并为未来的研究提供了一个有价值的方向.通过进一步的优化和验证,该模型有望在临床诊断中发挥更大的作用,为医生提供更准确、更可靠的辅助诊断工具,从而改善AD患者的治疗效果和生活质量.
利益冲突
无
参考文献
Alzheimer disease in China (2015-2050) estimated using the 1% population sampling survey in 2015
[J].
2020-2050年中国阿尔茨海默病患病情况预测研究
[J].
A novel end-to-end hybrid network for Alzheimer's disease detection using 3D CNN and 3D CLSTM[C]// 2020 IEEE 17th international symposium on biomedical imaging (ISBI)
Research progress on imaging classification of Alzheimer’s disease based on deep learning
[J].
基于深度学习的阿尔兹海默症影像学分类研究进展
[J].
DOI:10.11938/cjmr20223013
[本文引用: 1]

随着全球老龄化的加剧与深度学习的发展,基于深度学习的阿尔兹海默症(AD)影像学分类成为当前的一个研究热点.本文首先阐述了AD影像学分类任务中常用的深度学习模型、评估标准及公开数据集;接着讨论了不同图像模态在AD影像学分类中的应用;然后着重探讨了应用于AD影像学分类的深度学习模型改进方法;进一步引入了对模型可解释性研究的探讨;最后总结并比较了文中提及的分类模型,归纳了与AD影像分类相关的大脑区域,并对该领域未来的研究方向进行了展望.
Voxel-based morphometry: improving the diagnosis of Alzheimer’s disease based on an extreme learning machine method from the ADNI cohort
[J].
Tensor-based morphometry as a neuroimaging biomarker for Alzheimer's disease: an MRI study of 676 AD, MCI, and normal subjects
[J].
DOI:10.1016/j.neuroimage.2008.07.013
PMID:18691658
[本文引用: 1]

In one of the largest brain MRI studies to date, we used tensor-based morphometry (TBM) to create 3D maps of structural atrophy in 676 subjects with Alzheimer's disease (AD), mild cognitive impairment (MCI), and healthy elderly controls, scanned as part of the Alzheimer's Disease Neuroimaging Initiative (ADNI). Using inverse-consistent 3D non-linear elastic image registration, we warped 676 individual brain MRI volumes to a population mean geometric template. Jacobian determinant maps were created, revealing the 3D profile of local volumetric expansion and compression. We compared the anatomical distribution of atrophy in 165 AD patients (age: 75.6+/-7.6 years), 330 MCI subjects (74.8+/-7.5), and 181 controls (75.9+/-5.1). Brain atrophy in selected regions-of-interest was correlated with clinical measurements--the sum-of-boxes clinical dementia rating (CDR-SB), mini-mental state examination (MMSE), and the logical memory test scores - at voxel level followed by correction for multiple comparisons. Baseline temporal lobe atrophy correlated with current cognitive performance, future cognitive decline, and conversion from MCI to AD over the following year; it predicted future decline even in healthy subjects. Over half of the AD and MCI subjects carried the ApoE4 (apolipoprotein E4) gene, which increases risk for AD; they showed greater hippocampal and temporal lobe deficits than non-carriers. ApoE2 gene carriers--1/6 of the normal group--showed reduced ventricular expansion, suggesting a protective effect. As an automated image analysis technique, TBM reveals 3D correlations between neuroimaging markers, genes, and future clinical changes, and is highly efficient for large-scale MRI studies.
Inherent structure-based multiview learning with multitemplate feature representation for Alzheimer's disease diagnosis
[J].
Multimodal glioma segmentation with fusion of multiple self-attention and deformable convolutions
[J].
融合多重自注意力和可变形卷积的多模态脑胶质瘤分割
[J].
DOI:10.11938/cjmr20233059
[本文引用: 1]

脑胶质瘤的磁共振图像分割对于脑肿瘤的诊断、手术规划以及放疗等治疗方案的确定具有非常重要的意义.针对现有脑肿瘤分割算法分割精度不高边缘分割不精确,易出现假阳性的问题,本文提出一种基于多重自注意力和可变形卷积的Unet改进模型.模型将原始Unet框架的标准卷积替换为残差模块,以防止模型训练过程中出现梯度消失;通过在瓶颈层加入基于Transformer的多重自注意力模块来提取局部特征和全局上下文信息,以更好地挖掘像素间的相关性;在跨层连接处采用可变形卷积来增强模型对形状感知的敏感性,以提升肿瘤边缘特征的提取能力.实验结果表明,所提算法的分割结果评价指标高于使用同样数据集的其他对比模型,而且对肿瘤边缘的分割更加精确.这表明本文算法是一种有效的脑胶质瘤自动分割算法.
DMAF-Net: deformable multi-scale adaptive fusion network for dental structure detection with panoramic radiographs
[J].
Pfemed: Few-shot medical image classification using prior guided feature enhancement
[J].
Convolutional neural network based Alzheimer’s disease classification from magnetic resonance brain images
[J].
Multi-model and multi-slice ensemble learning architecture based on 2D convolutional neural networks for Alzheimer's disease diagnosis
[J].
Predicting Alzheimer's disease: a neuroimaging study with 3D convolutional neural networks
[J].
Recognition of Alzheimer's disease on sMRI based on 3D multi-scale CNN features and a gated recurrent fusion unit
[C]//
Construction of MRI-based Alzheimer’s disease score based on efficient 3D convolutional neural network: Comprehensive validation on 7,902 images from a multi-center dataset
[J].
An Alzheimer’s disease classification algorithm based on 3D-ResNet
[J].
基于3D-ResNet的阿尔兹海默症分类算法研究
[J].
CBAM: convolutional block attention module
[C]//
Deep residual learning for image recognition
[C]//
Identifying the regional substrates predictive of Alzheimer's disease progression through a convolutional neural network model and occlusion
[J].
DOI:10.1002/hbm.26026
PMID:35904092
[本文引用: 1]

Progressive brain atrophy is a key neuropathological hallmark of Alzheimer's disease (AD) dementia. However, atrophy patterns along the progression of AD dementia are diffuse and variable and are often missed by univariate methods. Consequently, identifying the major regional atrophy patterns underlying AD dementia progression is challenging. In the current study, we propose a method that evaluates the degree to which specific regional atrophy patterns are predictive of AD dementia progression, while holding all other atrophy changes constant using a total sample of 334 subjects. We first trained a dense convolutional neural network model to differentiate individuals with mild cognitive impairment (MCI) who progress to AD dementia versus those with a stable MCI diagnosis. Then, we retested the model multiple times, each time occluding different regions of interest (ROIs) from the model's testing set's input. We also validated this approach by occluding ROIs based on Braak's staging scheme. We found that the hippocampus, fusiform, and inferior temporal gyri were the strongest predictors of AD dementia progression, in agreement with established staging models. We also found that occlusion of limbic ROIs defined according to Braak stage III had the largest impact on the performance of the model. Our predictive model reveals the major regional patterns of atrophy predictive of AD dementia progression. These results highlight the potential for early diagnosis and stratification of individuals with prodromal AD dementia based on patterns of cortical atrophy, prior to interventional clinical trials.© 2022 The Authors. Human Brain Mapping published by Wiley Periodicals LLC.
Deep learning based multilevel classification of Alzheimer’s disease using MRI scans
[C]//
Development and validation of an interpretable deep learning framework for Alzheimer’s disease classification
[J].
Squeeze-and-excitation networks
[C]//
ECA-Net: Efficient channel attention for deep convolutional neural networks
[C]//
Diagnosis of Alzheimer’s disease via an attention-based multi-scale convolutional neural network
[J].
The Alzheimer's disease neuroimaging initiative (ADNI): MRI methods
[J].
DOI:10.1002/jmri.21049
PMID:18302232
[本文引用: 1]

The Alzheimer's Disease Neuroimaging Initiative (ADNI) is a longitudinal multisite observational study of healthy elders, mild cognitive impairment (MCI), and Alzheimer's disease. Magnetic resonance imaging (MRI), (18F)-fluorodeoxyglucose positron emission tomography (FDG PET), urine serum, and cerebrospinal fluid (CSF) biomarkers, as well as clinical/psychometric assessments are acquired at multiple time points. All data will be cross-linked and made available to the general scientific community. The purpose of this report is to describe the MRI methods employed in ADNI. The ADNI MRI core established specifications that guided protocol development. A major effort was devoted to evaluating 3D T(1)-weighted sequences for morphometric analyses. Several options for this sequence were optimized for the relevant manufacturer platforms and then compared in a reduced-scale clinical trial. The protocol selected for the ADNI study includes: back-to-back 3D magnetization prepared rapid gradient echo (MP-RAGE) scans; B(1)-calibration scans when applicable; and an axial proton density-T(2) dual contrast (i.e., echo) fast spin echo/turbo spin echo (FSE/TSE) for pathology detection. ADNI MRI methods seek to maximize scientific utility while minimizing the burden placed on participants. The approach taken in ADNI to standardization across sites and platforms of the MRI protocol, postacquisition corrections, and phantom-based monitoring of all scanners could be used as a model for other multisite trials.(c) 2008 Wiley-Liss, Inc.
FSL
[J].
DOI:10.1016/j.neuroimage.2011.09.015
PMID:21979382
[本文引用: 1]

FSL (the FMRIB Software Library) is a comprehensive library of analysis tools for functional, structural and diffusion MRI brain imaging data, written mainly by members of the Analysis Group, FMRIB, Oxford. For this NeuroImage special issue on "20 years of fMRI" we have been asked to write about the history, developments and current status of FSL. We also include some descriptions of parts of FSL that are not well covered in the existing literature. We hope that some of this content might be of interest to users of FSL, and also maybe to new research groups considering creating, releasing and supporting new software packages for brain image analysis.Copyright © 2011 Elsevier Inc. All rights reserved.
Grad-CAM: Visual explanations from deep networks via gradient-based localization
[C]//
Very deep convolutional networks for large-scale image recognition
[C]//
Densely connected convolutional networks
[C]//
An image is worth 16x16 words: Transformers for image recognition at scale
[J].
Research of spatial context convolutional neural networks for early diagnosis of Alzheimer’s disease
[J].